ESPiRiT을 이용한 Sensitivity Computation
Introduction
지난 글에서는 MRI의 개괄적인 원리, 즉 장비에서 얻은 raw data를 어떻게 이미지로 변환하는지에 대해 다루어보았습니다. 또한 그 중에서 scan time을 줄이기 위한 기법인 Parallel imaging과, 그로 인한 이미지 퀄리티 저하를 보상하는 알고리즘 중 SENSE, GRAPPA, 그리고 PRUNO에 대해 간략히 다루었습니다.
현재 가장 두루 쓰이는 방법은 GRAPPA, SENSE이지만 이 둘은 벌써 고안된 지 20년이 넘어가는 classic한 알고리즘입니다. Practical하진 않지만, 연구나 다른 특수한 목적으로 개발된 고성능 알고리즘들이 쏟아져나왔죠. 오늘은 그 중에서 수학적으로도 흥미롭고, 꽤 재미있는 인사이트를 주는 ESPiRiT (Uecker, 2014)의 원리와 의의, 구현법에 대해 알아봅니다.
Impact of Coil Sensitivity in MRI reconstruction
이 장의 이미지 출처는 Hamilton et al, 2017임을 밝힙니다.
MRI 장비에서는 인체 속 수소 원자가 방출하는 RadioFrequency Pulse, 즉 전자기 신호를 감지합니다. 이는 환자를 에워싸는 코일들에게 감지되는데, 당연히 코일마다 신호 source의 위치에 따라 감지하는 신호의 세기가 다릅니다.
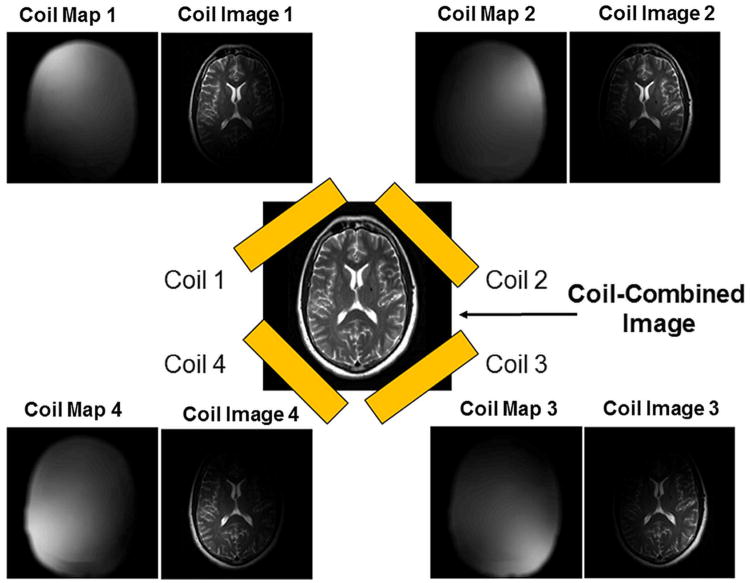
사람의 뇌에서 나오는 신호의 세기가 가운데 그림과 같다면, 각 코일에선 “Coil Sensitivity” (그림의 Coil Map) 라는 가중치가 곱해진 이미지를 얻게 됩니다. 대개 코일의 위치에서 가까운 부분의 신호를 더 잘 감지하는 것을 볼 수 있습니다.
가운데 사진과 같이 완전한 이미지를 얻기 위해서는 각 코일에서 얻은 이미지를 합성해야 합니다. 이 과정을 Coil-Combine이라고 하는데, 대개는 아래의 Sensitivity combine을 사용합니다.
\[m(\mathbf{r}) = \frac{\sum _ {c} S _ {c}(\mathbf{r})^{*} x _ {c}(\mathbf{r})}{\sum _ {c} \left\lvert S _ {c}(\mathbf{r}) \right\rvert^{2}}\]$S _ {c}(\mathbf{r})$은 $c$번째 코일의 위치 $\mathbf{r} = (x, y, z)$에서의 Sensitivity, $x _ {c}(\mathbf{r})$은 $c$번째 코일이 감지한 위치 $\mathbf{r}$의 신호.
Sensitivity를 알 수 없는 경우, 아래의 RSS (Root-Sum-Square) Combine을 대신 사용하기도 합니다.
\[m _ {\mathrm{RSS}}(\mathbf{r}) = \sqrt{\sum _ {c} \left\lvert x _ {c}(\mathbf{r}) \right\rvert^{2}}\]
두 방법은 $x _ {c}(\mathbf{r}) = S _ {c}(\mathbf{r})m(\mathbf{r})$, $\sum _ {c} \left\lvert S _ {c}(\mathbf{r})\right\rvert^{2} = 1$이 성립하는 ideal case에서 동등합니다. 하지만 $x _ {c}$에 noise가 있는 경우, $S$가 normalize되어 있지 않은 경우에는 Signal-to-noise ratio (SNR) 측면에서 전자가 더 좋은 방법입니다. 즉 Sensitivity를 알고 있는 경우에는 그렇지 않은 경우보다 질 높은 이미지를 만들 수 있는 것입니다. Coil combine과 같은 상황만이 아니라, Parallel imaging 등으로 이미지 퀄리티가 낮아진 상황에서도 Sensitivity Map은 큰 도움이 됩니다.
아래 두 식은 유명한 Parallel imaging technique에 Sensitivity map $S$가 사용되는 예시입니다.
SENSE (Pruessman, 1999) \(m = \left(S^{\mathrm{H}}\Psi^{-1} S + \lambda^{-1}\right)^{-1} S^{\mathrm{H}} \Psi^{-1}P\) $\ell _ {1}$-SPiRiT (Lustig, 2007) \(m = \arg\min _ {x} \left( \left\lVert y - \mathcal{UF}Sm \right\rVert _ {2}^{2} + \lambda \left\lVert \Psi m \right\rVert _ {1} \right)\)
Low frequency of Coil Sensitivity maps
Coil Sensitivity는 비교적 간단한 물리 법칙을 따르기 때문에, 공간에 따른 분포가 상당히 smooth하다는 점에서 계산이 꽤 용이하다는 장점이 있습니다. 다시 말해, 이를 k-space (Fourier domain) 상에서 나타냈을 때 high frequency 영역이 나타나지 않습니다.
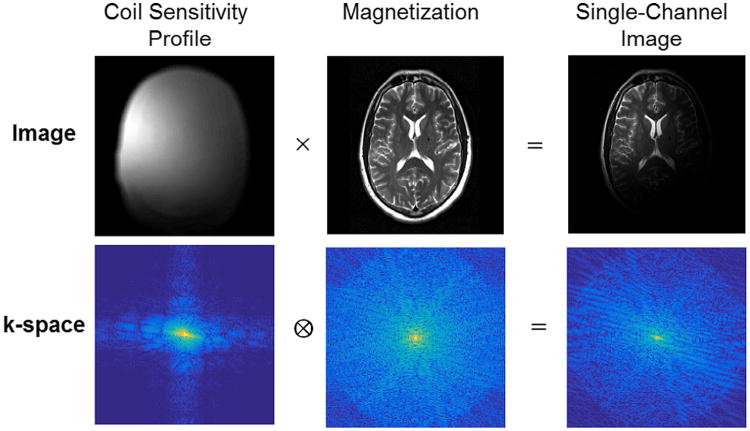
이미지 사이의 point-wise 곱은 k-space 상에서 convolution이 됩니다. 때문에 각 코일에서 얻어진 k-space signal은 “ground-truth k-space signal”에 coil sensitivity를 convolve한 것으로 볼 수 있는데, 많은 모형에서 coil-sensitivity를 small width convolution kernel로 가정합니다. 이미지 resolution이 256-1024인데 반해 보통 sensitivity convolution kernel의 크기는 10을 넘기지 않습니다.
Autocalibrating sensitivity maps
이미지만 보고 Sensitivity map을 알 수 있다면 좋겠지만, 그리 간단한 일이 아니기 때문에 보통은 Sensitivity map을 따로 측정하는 방법을 사용합니다. 하지만 이 과정에서 frame의 움직임, 코일의 측정 오류 등 여러 artifact가 누적되어 부정확한 결과를 가져올 수 있고, 때로는 Sensitivity 데이터를 사용하지 못하는 상황도 있습니다.
하지만 그리 간단하지 않은 일을 거의 처음으로 해내다시피 한 결과가 바로 오늘 리뷰할 논문인 ESPiRiT으로, 오직 k-space data를 사용하여 Sensitivity map을 eigenvector로 갖는 matrix $\mathcal{W}$를 설계하였습니다. 이렇게 얻으면 Sensitivity map을 별도로 측정하는 과정에서 생기는 여러 문제점을 회피할 수 있고, Sensitivity map을 사용하는 여러 고성능 Parallel imaging algorithm의 장점 또한 누릴 수 있습니다.
ESPiRiT – exploiting local correlation of k-space
다음과 같은 convention을 가정합시다.
- 이미지와 k-space는 모두 2차원. 다만 모든 논의는 3차원으로 자연스럽게 확장할 수 있습니다.
- Convolution kernel에서 중심점의 좌표는 항상 $\mathbf{0}$. width가 $2A+1$인 kernel $K$에 대해 $(K \ast X)(r) = \sum _ {x = -A}^{A} K(x)X(r - x)$ . (단, $\ast$ 는 convolution operator)
Local Linear Dependence of k-space
k-space위의 한 점 $\mathbf{k}$를 생각하고, $\mathbf{k}$ 를 중심으로 하는 한 변의 길이가 $w _ {d}$인 정사각형을 그려봅시다. 편의상 k-space가 2d이고 코일이 $C$개라고 가정하면, k-space data point는 총 $Cw _ {d}^{2}$개가 모일 것입니다. 각 코일의 Sensitivity map kernel의 크기가 $w _ {s}$라고 하면, 실제로 이 $Cw _ {d}^{2}$개의 점은 $(w _ {d} + w _ {s} - 1)^{2}$개의 magnetization data로 생성된 셈입니다. $C \gg 1$이므로 $w _ {d}$가 충분히 크다면, 이 점들 사이에 $\mathbf{k}$에 의존하지 않는 linear correlation이 생기게 될 것입니다. 이를 k-space의 local linear dependence라고 합니다.
GRAPPA (Griswold, 2002)는 이러한 linear correlation을 아주 잘 파고든 알고리즘입니다. 다음과 같은 $C \times C$ kernel $G$의 존재를 가정하는 것이죠. (여기서 $A \times B$ kernel이란 input channel이 $B$개이고 output channel이 $A$개인 convolution kernel을 의미하며 width가 $A \times B$라는 말과는 다릅니다)
- $G _ {c, d}(\mathbf{0}) = -\delta _ {cd}$
For k-space $X$, $G \ast X =0$ in every point of k-space.
- Width of $G$ (2 - 5) is way smaller than the width of $X$.
만약 parallel imaging으로 인해 어떤 point $\mathbf{r}$이 없어졌다면, Grappa kernel $G$의 도움을 받아 k-space point $X _ {c}(\mathbf{r})$을 복원할 수 있습니다.
\[0 = (G \ast X) _ {c}(\mathbf{r}) = -X _ {c}(\mathbf{r}) + \sum _ {\mathbf{s} \neq \mathbf{0}, d} G _ {cd}(\mathbf{s})X _ {d}(\mathbf{r - s})\]
이러한 $G$를 least-square method로 fitting하는 것이 grappa algorithm의 틀이라고 볼 수 있습니.
Fully Enjoying the null-space of $X$
ESPiRiT이나 PRUNO와 같은 method들은 GRAPPA에 비해 local linear correlation을 보다 유연하게 활용합니다. 이를 위해, “local linear correlation”을 보다 정확하게 정의해봅시다.
k-space의 각 점 $\mathbf{k}$에 대해, $\mathbf{k}$를 중심으로 $w _ {d} \times w _ {d}$ 정사각형 내에 위치한 $Cw _ {d}^{2}$개의 점들을 하나의 row vector로 나타냅시다. Local Linear Correlation을 구할 sample point가 $B$개 존재한다고 하면, 우리는 거대한 $B \times Cw _ {d}^{2}$ 크기의 tall matrix를 얻을 수 있습니다. 이렇게 만든 행렬을 block hankel matrix라고 부릅니다.
block hankel matrix $A$의 입장에서 local linear correlation이 있다는 말은 곧 $A$의 column rank가 낮다는 것을 의미합니다. 따라서 $A$를 span space와 null space로 구분하면 모든 correlation을 얻을 수 있을 것입니다. 이는 보통 $A$에 SVD(Singular Value Decomposition)를 적용하여 cutoff singular value $\sigma _ {0}$보다 singular value가 큰 singular vector를 span space로, 작은 singular vector를 null-space로 보내는 것으로 구분합니다.
Formulating flexibilized local correlations
$A$의 singular vector 중 span space로 분류된 것을 $V _ {\Vert}$, null space로 분류된 것을 $V _ {\perp}$라고 둡시다. $V _ {\perp}$는 hankel matrix에 대한 eigenvector(정확히는 singular vector)로도 볼 수 있지만, Hankel matrix의 정의를 생각해보면 그 본질은 convolution kernel입니다. $V _ {\perp}$의 matrix size가 $Cw _ {d}^{2} \times U$라고 두면, $V _ {\perp}$를 $U \times C$ convolution kernel로 볼 수 있는 것입니다. Nulling kernel의 본질에 따라 $V _ {\perp} \ast X = 0$이라는 convolution-style의 관계식이 얻어지지만, concise한 식 전개를 위해 조금 더 hankel matrix 위에서 논리를 전개해봅시다.
$X$를 길이가 $CN _ {x}N _ {y}$인 거대한 벡터로 생각하는 대신 “Extraction operator” $R _ {\mathbf{k}}$를 정의하여, $R _ {\mathbf{k}}X$가 $\mathbf{k}$를 중심으로 하는 길이 $Cw _ {d}^{2}$짜리 벡터가 되도록 합시다. 즉, 기존에 만든 hankel matrix $A$는 Sampling location이 $\mathbf{k} _ {1}, \cdots, \mathbf{k} _ {B}$라면 $A = \begin{bmatrix} R _ {\mathbf{k} _ {1}} X & \cdots & R _ {\mathbf{k} _ {B}} X \end{bmatrix}^{\mathrm{H}}$가 되는 셈입니다. 이 notation을 도입하면
$V _ {\perp} \ast X = 0 \implies V _ {\perp}^{\mathrm{H}} R _ {\mathbf{k}}X = 0 \;\forall \mathbf{k}.$
로 linear-algebra style 관계식을 쓸 수 있습니다. 좌변의 $V _ {\perp}$는 kernel, 우변의 $V _ {\perp}$는 matrix임에 주의하세요. 위 식의 우변을 positive semidefinite form으로 적고, 모든 $\mathbf{k}$에 대해 더하면 아래와 같은 식을 얻습니다.
\[\sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}} V _ {\perp} V _ {\perp}^{\mathrm{H}} R _ {\mathbf{k}} X = 0.\]SVD의 orthonormality에 따라 $V _ {\perp}V _ {\perp}^{\mathrm{H}} + V _ {\Vert}V _ {\Vert}^{\mathrm{H}} = I$이므로, \(\sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}}(V _ {\Vert}V _ {\Vert}^{\mathrm{H}})R _ {\mathbf{k}} X = \sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}}R _ {\mathbf{k}} X\) 이 때 $\sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}}R _ {\mathbf{k}}$는 정확히 $Cw _ {d}^{2} \cdot I$가 됨을 알 수 있습니다. (with periodic boundary condition on k-space) $M = Cw _ {d}^{2}$로 두면, 아래 관계식이 유도됩니다. \(\left(\frac{1}{M}\sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}}V _ {\Vert}V _ {\Vert}^{\mathrm{H}}R _ {\mathbf{k}}\right) X = X\) 이 때 좌변에 가해진 operator $\left(\frac{1}{M}\sum _ {\mathbf{k}} R _ {\mathbf{k}}^{\mathrm{H}}V _ {\Vert}V _ {\Vert}^{\mathrm{H}}R _ {\mathbf{k}}\right)$를 $\mathcal{W}$라고 두면, $\mathcal{W}X = X$를 만족하므로 $X$는 $\mathcal{W}$의 eigenvector (with eigenvalue $1$)이 됨을 알 수 있습니다.
The “ESPiRiT operator” $\mathcal{W}$
$\mathcal{W}$를 보고 첫눈에 알아챌 수 있는 성질은 $\mathcal{W}$가 Positive semidefinite, hermitian operator라는 것입니다. 따라서 모르긴 몰라도 Eigenvector를 구하는 과정이 쉬울 것 같습니다.
차근차근 뜯어보면 알 수 있는 성질은 $\mathcal{W}$의 eigenvalue가 $1$ 이하라는 것입니다. SVD의 orthonormality에 의해 $V _ {\Vert}V _ {\Vert}^{\mathrm{H}}$가 projection operator가 되어 eigenvalue가 1이하가 되고, 한 k-space position $\mathbf{k}$에 대해 정확히 $M$개의 summand가 영향을 주기 때문입니다. 따라서 $X$는 단순히 “특정 고윳값을 갖는 eigenvector” 정도가 아니라 “maximum eigenvector”가 됩니다.
$V _ {\perp}$ (나아가 $V _ {\Vert}$)를 convolution operator로 볼 수 있듯이, $\mathcal{W}$ 또한 convolution kernel로 볼 수 있습니다. 구체적으로 $\mathcal{W}$는 $C \times C$ kernel이 되는데, 직관적으로 알 수 있는 사실이니 식을 찬찬히 들여다보고 유도해보시기 바랍니다.
Key theorem: Point-wise decomposition of $\mathcal{W}$
MRI에서 가장 중요한 식 중 하나인 Signal equation을 복기해보면 다음과 같습니다.
$X _ {c}(\mathbf{k}) = \sum _ {\mathbf{r}} \omega^{-\mathbf{r} \cdot \mathbf{k}} S _ {c}(\mathbf{r})m(\mathbf{r}) = \mathcal{F} \left\lbrace S _ {c} \odot m \right\rbrace(\mathbf{k})$
$\odot$은 element-wise product를 matrix multiplication과 구분하기 위한 표기이고, $\mathcal{F}$는 discrete fourier transform입니다. 이 식을 Eigenvalue equation $\mathcal{W}X = X$에 끼워넣으면 \(\left(\mathcal{F}^{-1}\mathcal{W}\mathcal{F}\right)(S _ {c} \odot m) = S _ {c} \odot m\) 이 됩니다. 따라서 Operator $\mathcal{G} = \mathcal{F}^{-1}\mathcal{W}\mathcal{F}$이 상당히 궁금해지는데, 꽤 비직관적인 아래 사실이 성립합니다.
$\mathcal W$는 convolution이기때문에 $\tilde{\mathcal W} := \mathcal F^{-1} \mathcal W \mathcal F$ 는 pointwise matrix operation으로 분리 된다: $\tilde{\mathcal W}\big\vert _ {q} = \mathcal G _ q = G _ q^H G _ q$.
이것이 사실이라고 한다면, $\mathcal{G} _ {q}$는 점을 고정한 채 convolution channel끼리만 mixing하는 $C \times C$ positive semidefinite matrix가 되고, $\mathcal{G}$는 batch matrix multiplication이 됩니다. 따라서 거대한 크기의 matrix $\mathcal{W}$와 씨름할 필요 없이 각 점 $\mathbf{q}$마다 $\mathcal{G} _ {q}S _ {c}(\mathbf{q})m(\mathbf{q}) = S _ {c}(\mathbf{q})m(\mathbf{q})$를 해결하면 되고, $m(\mathbf{q}) \neq 0$ 조건에서 길이 $C$짜리 벡터 $S _ {\cdot}(\mathbf{q})$는 $\mathcal{G} _ {q}$의 maximum eigenvector가 됩니다.
Proof of Key theorem
결국 다음과 같은 명제를 증명하면 됩니다.
$K$가 $C \times C$ convolution이면, $\mathcal{F}^{-1}K\mathcal{F}$는 batch of $C \times C$ matrix multiplication이 된다.
$K$가 positive semidefinite이라면 $\mathcal{F}^{-1}K\mathcal{F}$ 역시 PSD인 것은 자명하기 때문에 $\mathcal{G} _ {q} = G _ {q}^{\mathrm{H}}G _ {q}$는 추가로 증명할 필요가 없습니다.
$Y = K \ast X$라는 convolution 관계식에서 $Y = \mathcal{F}y, X = \mathcal{F}x, K = \mathcal{F}L$이라고 두면 우리는 $\mathcal{F}^{-1}K\mathcal{F}x = y$가 $x, L$에 대한 식으로 어떻게 쓰이는지 보면 됩니다. \(\begin{aligned} y _ {c}(\mathbf{r}) &= \sum _ {\mathbf{k}} Y _ {c}(\mathbf{k}) \omega^{\mathbf{r} \cdot \mathbf{k}}\\ &= \sum _ {\mathbf{k,l},d} K _ {cd}(\mathbf{l})X _ {d}(\mathbf{k - l})\omega^{\mathbf{r} \cdot \mathbf{k}}\\ &= \sum _ {\mathbf{k, l, u, v}, d} L _ {cd}(\mathbf{u})x _ {d}(\mathbf{v}) \omega^{\mathbf{r \cdot k -u \cdot l - v \cdot (k - l)}}\\ &= \sum _ {\mathbf{u,v},d} L _ {cd}(\mathbf{u})x _ {d}(\mathbf{v}) \delta _ {\mathbf{r, v}}\delta _ {\mathbf{r, u}}\\ &= \sum _ {d} L _ {cd}(\mathbf{r})x _ {d}(\mathbf{r})\quad \square \end{aligned}\)
Exercise. $G _ {q}$가 사실은 $V _ {\Vert}$의 Fourier transform이 됩니다. $\mathcal{W}$의 식으로부터 이를 유도하세요.
Computation of ESPiRiT
위의 복잡한 과정으로 Sensitivity map을 구하고 나면, 구한 Sensitivity map을 SENSE, SPiRiT 등의 알고리즘에 꽂아넣는 것으로 undersampled k-space를 채울 수 있습니다. SPiRiT은 일종의 Compressive Sensing 알고리즘으로, 이 글에서 대략적인 원리를 공부할 수 있습니다.
ESPiRiT은 BART 그룹의 리더 Uecker가 만든 알고리즘이다보니, 저자가 구현한 C 기반 코드가 깃허브에 공개되어 있습니다. Sensitivity map을 만들기 위한 computational task를 간단히 아래와 같이 나눌 수 있습니다.
- Block Hankel matrix $A$를 만들고, $A$의 SVD로 부터 $V _ {\Vert}$와 $V _ {\perp}$를 만들기.
- $V _ {\Vert}$의 Fourier transform으로 $G _ {q}$를 만들기
- $G _ {q}$ ($C \times C$행렬, 총 $N _ {x}N _ {y}$개의 batch) matrix의 maximum singular vector 찾기.
- 이미지 퀄리티를 위해 maximum singular vector가 아닌, 가장 큰 $k \approx 4$개의 maximum singular vector를 찾아야 하는 경우가 있음.
이 중 첫 번째 SVD는 그다지 시간을 요구하지 않습니다. 다만 $G _ {q}$ matrix가 최대 $C \times (Cw _ {d}^{2} - U) \times N _ {x}N _ {y}$ 까지 커질 수 있고, $\mathcal{G} _ {q} = G _ {q}^{H}G _ {q}$가 $C \times C \times N _ {x}N _ {y}$ 크기의 batch of matrices가 되는 것이 부담이라면 부담입니다. 또한 각 행렬마다 $\mathcal{O}(C^{3})$ 시간이 소요되는 SVD를 사용하는 것이 많은 시간을 소모하기도 합니다. 이를 위한 좋은 대책은
CUDA기반의 라이브러리로 빠른 batch svd를 사용하기.- 가장 큰 $k$개의 eigenvector를 찾기 위해 SVD 대신 Power iteration (혹은 그 일반화인 Orthogonal iteration) 구현하기
등이 있습니다. BART는 수 년간 코드베이스를 관리하며 이러한 최적화들을 적용해왔지만, 제가 확인했을 때 아직 최적화할 부분이 많이 남아있습니다. C나 CUDA를 이용한 오픈 소스 contribution을 원하시고, 이러한 task에 관심이 있다면 연락 주시기 바랍니다.
Further Reading
SENSE, GRAPPA는 무난한 알고리즘이지만 각각 pitfall을 가지고 있습니다. SENSE의 경우 object의 크기가 너무 커 field-of-view를 넘어가는 경우 기존의 알고리즘으로 reconstruction이 어렵고, GRAPPA는 acceleration factor가 높거나 noise가 많이 낀 데이터에서 급격한 성능 저하를 보입니다. ESPiRiT에서 여러 개의 maximum eigenvector를 얻고 Soft-SENSE 기법으로 reconstruction을 진행하면 이를 모두 회피할 수 있는데, 자세한 부분은 ESPiRiT 논문과 Reference에 걸린 링크를 참고하시면 좋습니다.
ESPiRiT 이외에 Sensitivity estimation을 시도한 논문은 JSENSE (Ying, 2007)이 있습니다. ESPiRiT보다는 훨씬 직관적인 polynomial fitting algorithm인데, 실제로 사용되는 것을 많이 보지는 못했습니다.
Reference
ESPiRiT: Uecker, M., Lai, P., Murphy, M. J., Virtue, P., Elad, M., Pauly, J. M., Vasanawala, S. S., & Lustig, M. (2014). ESPIRiT–an eigenvalue approach to autocalibrating parallel MRI: where SENSE meets GRAPPA. Magnetic resonance in medicine, 71(3), 990–1001. https://doi.org/10.1002/mrm.24751
SENSE: Pruessmann KP, Weiger M, Scheidegger MB, Boesiger P. SENSE: sensitivity encoding for fast MRI. Magn Reson Med. 1999 Nov;42(5):952-62. PMID: 10542355.
GRAPPA: Griswold MA, Jakob PM, Heidemann RM, Nittka M, Jellus V, Wang J, Kiefer B, Haase A. Generalized autocalibrating partially parallel acquisitions (GRAPPA). Magn Reson Med. 2002 Jun;47(6):1202-10. doi: 10.1002/mrm.10171. PMID: 12111967.
JSENSE: Ying L, Sheng J. Joint image reconstruction and sensitivity estimation in SENSE (JSENSE). Magn Reson Med. 2007 Jun;57(6):1196-202. doi: 10.1002/mrm.21245. PMID: 17534910.
SENSE의 결점: FOV가 작을 때: Griswold MA, Kannengiesser S, Heidemann RM, Wang J, Jakob PM. Field-of-view limitations in parallel imaging. Magn Reson Med. 2004 Nov;52(5):1118-26. doi: 10.1002/mrm.20249. PMID: 15508164.